ORFanID -En online søkemotor for å identifisere orfangener
{Innhold i parentes {} og understrekninger er ved oversetter.}
Av Jonathan McLatchie, 13. nov. 2024. Oversatt herfra
 Taksonomisk begrensede gener er {protein} kodende sekvenser som er begrenset til en spesifikk taksonomisk gruppe og mangler gjenkjennelige homologer i andre grupper, inkludert nært beslektede arter. Eksistensen av slike gener er over-raskende. gitt hypotesen om felles universell avstamning - dvs. at alle gener har utviklet seg ved mange påfølgende modifikasjoner fra andre gener, og sporet helt tilbake til den siste universelle felles stamfaren. Noen gener, kalt orfan -gener, er unike og finnes bare i en enkelt art.
Taksonomisk begrensede gener er {protein} kodende sekvenser som er begrenset til en spesifikk taksonomisk gruppe og mangler gjenkjennelige homologer i andre grupper, inkludert nært beslektede arter. Eksistensen av slike gener er over-raskende. gitt hypotesen om felles universell avstamning - dvs. at alle gener har utviklet seg ved mange påfølgende modifikasjoner fra andre gener, og sporet helt tilbake til den siste universelle felles stamfaren. Noen gener, kalt orfan -gener, er unike og finnes bare i en enkelt art.
Intelligente designteoretikere har lenge vært interessert i orfangener og Taksonomisk Gegrensede Gener (TBG). Hvis det ikke eksisterer andre lignende gen som et bestemt gen, fra hvilken genkilde utviklet de seg? I fjor ble et Discovery Institute-finansiert forskningsoppgave publisert i tidsskriftet PLoS One, og introduserte en "grafisk nettbasert søkemotor", kalt Orfanid, som "letter effektiv identifisering av både foreldreløse {Orfan} gener og TBG-ere på alle taksonomiske nivåer, fra DNA- eller aminosyresekvenser i NCBI -databaseklyngen og andre store bioinformatikk -depoter. ”{1) Programvaren gjør det mulig for brukere å" identifisere gener som er unike for enhver taksonomisk rang, fra arter til domene, ved bruk av NCBI -systematiske klassifiserere. "
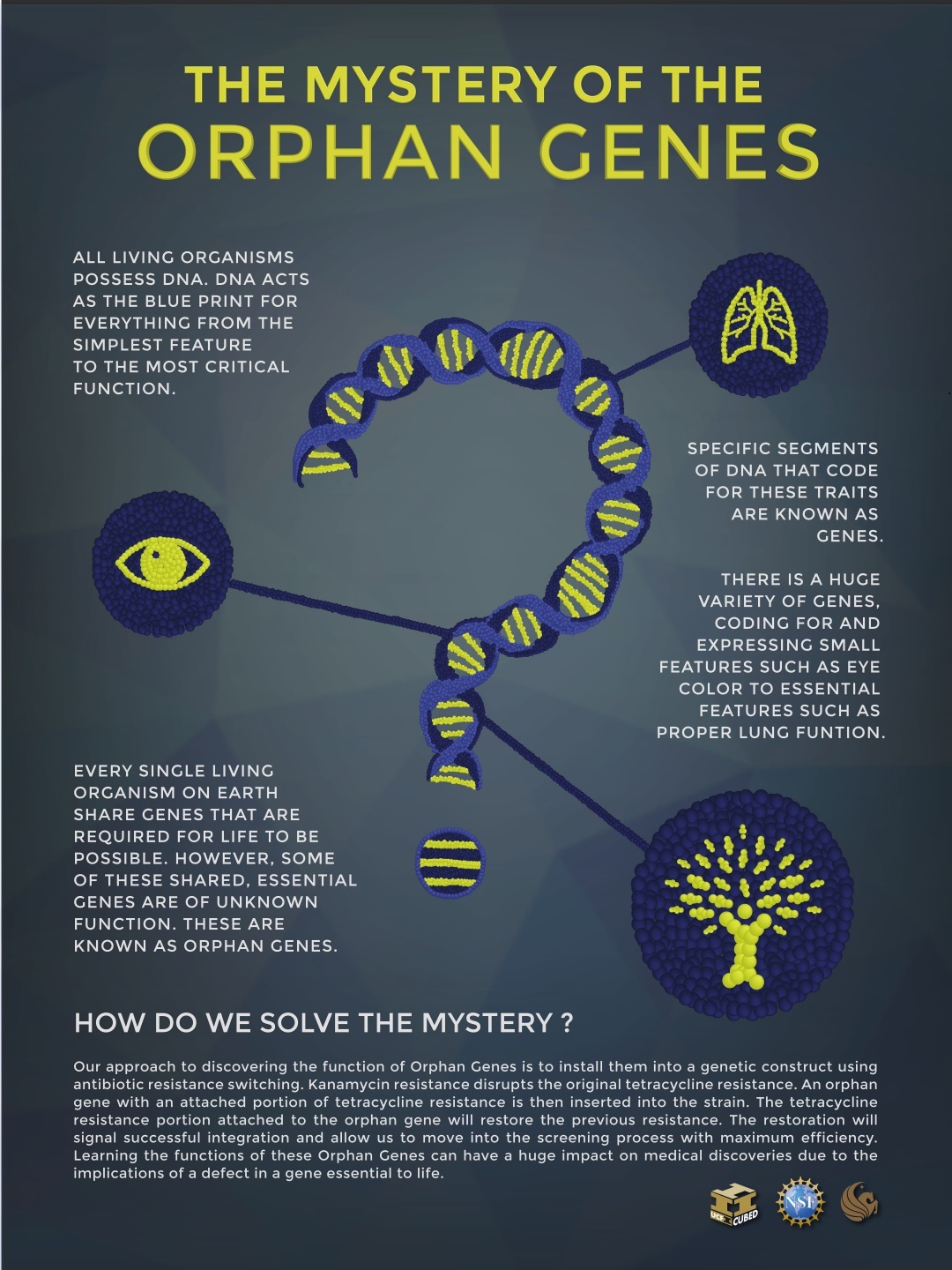
Bilde 1. DNA
Begrensninger i andre programmer
Forfatterne bemerker begrensningene til andre tilgjengelige genomiske analyseprogrammer som man kan bruke for å studere foreldreløse gener:
For øyeblikket kan verktøyalternativene forskere finner i dag for å studere foreldreløse gener begrenset, ettersom de fleste programvareløsninger fokuserer på å identifisere ortologer eller slutte til Ortho -grupper og generelt, er begrenset til proteiner. For eksempel er Orfanfinder -funksjonalitet begrenset til planter, bakterier og sopp, og URLen i den opprinnelige publikasjonen (DOI: 10.1093/bioinformatikk/BTW122) er ikke aktiv. Imidlertid har et websøk avslørt at Orfanfinder er tilgjengelig her, selv om denne programvaren ikke har blitt oppdatert siden 2016. Sequencever utfører BLAST uten å klassifisere proteiner/DNA -sekvenser til taksonomiske nivåer.
Programvaren Geneious kan utføre justering og bygge et fylogenetisk tre, men å identifisere foreldreløse ved bruk av denne programvaren er utfordrende. Tilsvarende gir OrthoFinder muligheten til å bruke DIAMOND eller den anbefalte MMSEQ2 for sekvensjustering. OMA Orthology eller serien med analytiske ressurser utviklet av Bioinformatics Resource Centers (BRCS) for smittsomme sykdomsprogram [Bioinformatics Tools, WorkSpaces and Services for Bioinformatics Data Analysis som Amoebadb, Fungidb, Orthomcl] viser bare ortologe gener/proteiner og identifiserer ikke Foreldreløse gener. En nyere genklassifiserings-plattform, www.shoot.bio, kan også være nyttig for å justere og sammenligne genopprinnelse, men den bruker bare protein sekvenser. Kort sagt, disse verktøyene utfører justering eller identifiserer konserverte gener fra genomer, men de er ikke egnet til å identifisere foreldreløse {ORFan} gener slik ORFanID designet til å gjøre. [Interne siteringer utelatt.]
De fremhever videre særegenheten til ORFanID i forhold til andre tilgjengelige genomiske analyseprogrammer:
Orfanids egenart er i tre aspekter: 1) den behandler ikke bare protein/aminosyresekvenser, men også DNA/nukleotidsekvenser. 2) Med sin innebygde homologitolk og klassifiserer gir denne søkemotoren den taksonomiske rangering av et gen enten som et foreldreløst gen eller som et gen begrenset til et taksonomisk nivå i livets tre; 3) Når orfans og TRG -er blir identifisert, bygger Orfanid sin egen database med resultatene av analysen og gir forskeren muligheten til å utforske dataene ytterligere.
Testing av programmet
Forfatterne testet bruken av programmet sitt ved å bruke det til å analysere DNA og proteinsekvenser av organismer inkludert C. elegans, S. cerevisiae, D. melanogaster og H. sapiens, ved bruk av gener som tidligere hadde blitt spådd å være taksonomisk begrenset. Resultatene deres bekreftet "at orfanid-algoritmen er nøyaktig når det gjelder å klassifisere proteiner til deres respektive taksonomiske gruppe, basert på funksjonaliteten til BLAST og valg av parameter-innstillinger." De testet deretter programmet på artsspesifikke gener {dvs. foreldreløse), ved bruk av gener som tidligere hadde blitt identifisert som foreldreløse i A thaliana, D. melanogaster, C. elegans og S. cerevisiae. Resultatet var at "ORFanID nøyaktig identifiserte disse publiserte genene som artsspesifikke foreldreløse {gener)." Forfatterne konkluderte med at "samlet, antyder disse resultatene at orfanid fungerer nøyaktig og pålitelig med å klassifisere og identifisere artsspesifikke foreldreløse gener."
Bilde 2. Bok om såkalte foreldreløse gener
Forfatterne sammenlignet også følsomheten og nøyaktigheten til ORFanID, i riktig klassifisering av taksonomisk begrensede gener ved bruk av fem organismer (C. elegans, E. coli, H. sapiens, O. sativa og Z. mays). Resultatene avslørte at "ORFanID er mer følsom og gir mer nøyaktige resultater i klassifisering av foreldreløse og strikte orfangener."
Selv om det en gang var antatt at orfangener var sjeldne, har forskning avslørt at taksonomisk begrensede gener (og til og med strikte orfan -gener, som er begrenset til en enkelt art) faktisk er ganske vanlige. ORFanID er et verktøy som kan brukes i videre forskningsprosjekter for å utforske og katalogisere taksonomisk begrensede gener og forbedre vår forståelse av deres utbredelse. Opprinnelsen og naturen til orfangener er forskningsspørsmål som alle er interessert i - enten de gjør vitenskap fra et evolusjonært paradigme eller intelligent design. Dette prosjektet viser at ID er med på å inspirere forskning som er av interesse for vitenskap generelt og bærer god frukt for det vitenskapelige samfunnet.
Referanser
1. Gunasekera RS, Raja KKB, Hewapathirana S, Tundrea E, Gunasekera V, Galbadage T, Nelson PA. ORFanID: A web-based search engine for the discovery and identification of orphan and taxonomically restricted genes. PLoS One. 2023 Oct 25;18(10):e0291260. doi: 10.1371/journal.pone.0291260. Erratum in: PLoS One. 2024 Aug 8;19(8):e0308834. doi: 10.1371/journal.pone.0308834. PMID: 37879070; PMCID: PMC10599687.
Oversettelse, via google oversetter, og bilder ved Asbjørn E. Lund
 Taksonomisk begrensede gener er {protein} kodende sekvenser som er begrenset til en spesifikk taksonomisk gruppe og mangler gjenkjennelige homologer i andre grupper, inkludert nært beslektede arter. Eksistensen av slike gener er over-raskende. gitt hypotesen om felles universell avstamning - dvs. at alle gener har utviklet seg ved mange påfølgende modifikasjoner fra andre gener, og sporet helt tilbake til den siste universelle felles stamfaren. Noen gener, kalt orfan -gener, er unike og finnes bare i en enkelt art.
Taksonomisk begrensede gener er {protein} kodende sekvenser som er begrenset til en spesifikk taksonomisk gruppe og mangler gjenkjennelige homologer i andre grupper, inkludert nært beslektede arter. Eksistensen av slike gener er over-raskende. gitt hypotesen om felles universell avstamning - dvs. at alle gener har utviklet seg ved mange påfølgende modifikasjoner fra andre gener, og sporet helt tilbake til den siste universelle felles stamfaren. Noen gener, kalt orfan -gener, er unike og finnes bare i en enkelt art.